▼更多精彩推荐,请关注我们▼文章信息
标题:CompleteStereoinversion of L?Tryptophanby a Fungal SingleModule Nonribosomal Peptide Synthetase
DOI:10.1021/jacs.9b08898
接收时间:2019.08.17
发表时间: 2019.10.01
期刊:JACS
影响因子:14.695文章作者

唐奕
武汉大学药学院讲座教授、博士生导师,美国加州大学洛杉矶分校(UCLA)化学与生物分子工程系和化学与生物化学系教授。
研究方向:
真菌次级代谢产物的生物合成与关键酶的催化机制
西澳大利亚大学(UWA)Yit Heng Chooi教授,日本静冈県立大学的Kenji Watanabe教授,北微所尹文兵研究员,高书山研究员,北京协和医学院胡友财教授,西南大学邹懿教授,上海交大唐满成教授,中国海洋大学李德海教授曾在其实验室学习和工作。
个人主页:https://www.chemistry.ucla.edu/directory/tang-yi
▍研究背景
单模块 NRPS 和 NRPS-like 酶采用类似的硫化模板酶学,具有一组腺基化结构域(A domain)和硫化结构域(T domain)。羧酸被 A 结构域活化之后,底物在 T 结构域上被保存为磷酸戊硫酸酯(Ppant)。根据下游释放结构域的类型,硫代酯中间体可进行广泛的修饰,例如:
A-T-C 模块中的缩合结构域(C-domain)参与酯化/酰胺化反应;
A-T-TE 模块中的硫酯酶结构域(TE domain)参与 Dieckman/aldol 缩合或环化反应;
A-T-R 或A-T-R-R 中的还原酶结构域(R domain)进行二电子或四电子还原;
A-T-R-P 模块中通过 PLP 依赖的 Aldol 缩合参与了双电子还原反应。
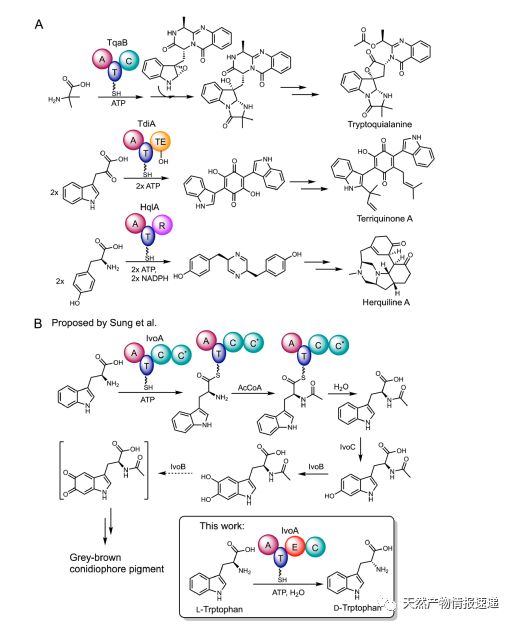
图1.单模块NRPS和NRPS-like酶的不同功能。(A)已鉴定的例子;(B)本文中的IvoA
一般认为酶催化产物 N-乙酰-L-色氨酸被 P450 酶 IvoC 和酚氧化酶 IvoB 进一步氧化,从而参与到孢子色素的合成路线中。其中,ivoA 的乙酰转移酶活性不太可能适合单模块NRPS合成酶。原因如下:
(1)通过乙酰辅酶A实现N-乙酰化,以1当量ATP为代价激活底物的羧基是代谢浪费。
(2)下游C结构域以上游T结构域为受体进行缩合反应(这里为乙酰化)是违反NRPS方向性规则的。
▍研究内容
美国加利福尼亚大学洛杉矶分校的唐奕课题组最近在JACS期刊发表了题为“Complete Stereoinversion of L?Tryptophan by a Fungal Single-ModuleNonribosomal Peptide Synthetase”的研究论文,该研究证明单模块 NRPS 合成酶 IvoA 催化 L-色氨酸通过 ATP 依赖性单向立体转化完全转化为 D-色氨酸。虽然立体转化是由差向异构化(E)结构域催化的,但末端缩合(C)结构域立体选择性地水解 D-tryptophanyl-S-phasphapantetheine 的硫酯键。
为了分析 IvoA 酶的功能,作者首先分析了 ivoA 的域结构,由于异构化结构域(E domain)与末端缩合结构域(C domain)结构域具有序列和结构同源性,并且经常插入到 NRPS 装配线的 T domain 和 C domain 之间,因此作者假设 IvoA 的真正的域结构是 A-T-E-C。
为了验证这一假说,作者首先在 Saccharomyces cerevisiae JHY686 中过表达 IvoA,通过手性 HPLC 分析发现 N-乙酰化- L-色氨酸已全部转化为 N-乙酰化-D-色氨酸(图2A),色氨酸的立体化学转化证明在 IvoA 中存在一个 E domain。
随后在通过体外酶活实验证明 IvoA 的功能,IvoA 酶 A domain的底物特异性实验证明 L-色氨酸是最优底物,用 LC-MS 监测在重水中的反应,发现了色氨酸的同位素峰(图2B),氢谱分析发现氢-氘交换发生在Cα-H(图2C),用手性 HPLC 监测反应,发现 L-色氨酸只需要 3 个小时就可以完全转化为 D-色氨酸(图2D)。整个酶促动力学实验说明 E domain和 C domain对 IvoA 的催化功能至关重要,当失活 E domain 时,kcat下降了 420 倍,失活 C domain 时,kcat 下降了 4700 倍。以上实验表明 IvoA 在体外缺乏乙酰基转移酶活性,但它是一种真正的 ATP 依赖性酶,能够催化 L-色氨酸立体转化为 D-色氨酸,D-色氨酸在体内的乙酰化必须通过内源乙酰基转移酶进行。
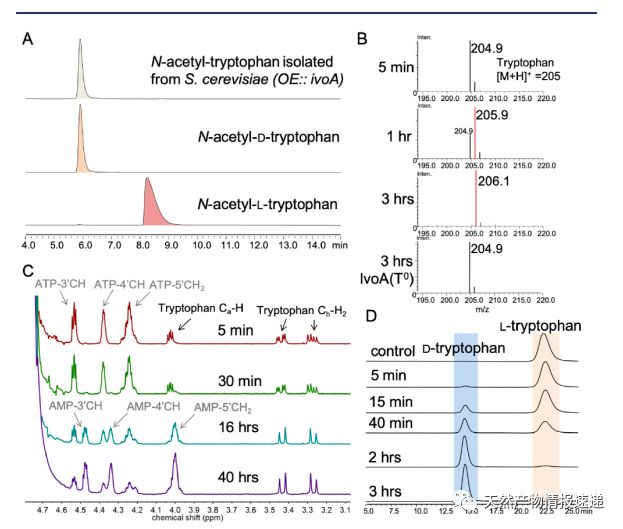
图 2. IvoA活性的表征
与常见的依赖于或不依赖于 PLP 的氨基酸消旋酶不同(方案1),IvoA 的作用机制是将色氨酸作为 Tryptophanyl-S-Ppant 硫酯传递到 E domain,通过异构化反应得到 D/L-Tryptophanyl-S-Ppant 的非对映异构体。C domain 通过水解 D-Tryptophanyl-S-Ppant 硫代酯完成动力学拆分(图3)。然而,IvoA 不能将 D-tryptophan 转化为 L-Tryptophan,这表明 C domain 不能水解 L-tryptophanyl-S-Ppant。
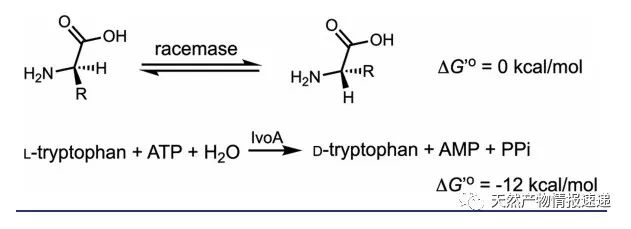
方案 1. 用普通外消旋酶和 IvoA 将氨基酸对映体立体转化为镜像对映体
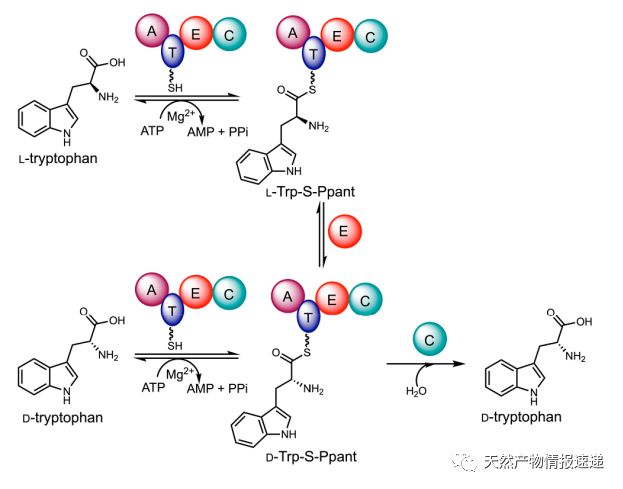
图 3. IvoA 的作用机制
为了验证 IvoA 中 C domain的立体选择性,作者将纯化出的 IvoA-C 添加到IvoA-ΔC 突变菌株中,发现 L-tryptophanyl 成功转化为 D-tryptophanyl,证明了 IvoA-C 的立体选择性(图4)。将 L/D-tryptophanyl 加入到 IvoA-ΔC-ΔE 突变株中,通过蛋白质质谱发现了 D/L-tryptophanyl-S-Ppant。当除去过量的 D/L-tryptophan 底物时,IvoA-C 开始立体选择性催化 D-tryptophanyl-S-Ppant。

图4. IvoA中C domain的活性表征
▍研究总结
综上所述,作者对IvoA的生物化学研究发现了非同寻常的活性,扩展了单模块NRPS的功能多样性。对IvoA功能的重新鉴定也让我们对真菌色素的生物合成有了更深刻的了解。
撰文:李鸿成
责编:李鸿成


点击“阅读原文”,即可下载文章